简介
这篇文章介绍了降维的基本思路及用奇异值分解(SVD)进行主成分分析(PCA)的操作。
依赖
import numpy as np
from numpy.linalg import svd
import matplotlib
%matplotlib inline
import matplotlib.pyplot as plt
from sklearn.datasets import make_swiss_roll
from sklearn.manifold import LocallyLinearEmbedding
from sklearn.decomposition import PCA降维的基本思路
投射
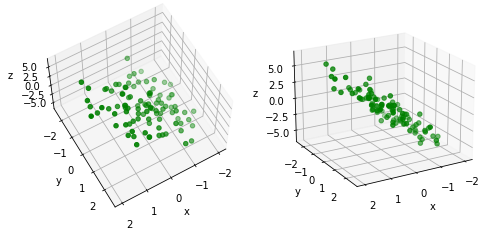
如果数据点不是在各个维度上均匀地分布着:
N = 100
x = np.random.normal(0, 1, N)
y = np.random.normal(0, 1, N)
z = 3 * x + np.random.normal(0, 1, N)
fig = plt.figure(figsize=(8,8))
ax1 = fig.add_subplot(1, 2, 1, projection='3d')
ax1.scatter(x, y, z, c='g', marker='o')
ax1.set_xlabel('x')
ax1.set_ylabel('y')
ax1.set_zlabel('z')
ax1.view_init(60, 60)
ax2 = fig.add_subplot(1, 2, 2, projection='3d')
ax2.scatter(x, y, z, c='g', marker='o')
ax2.set_xlabel('x')
ax2.set_ylabel('y')
ax2.set_zlabel('z')
ax2.view_init(20, 60)
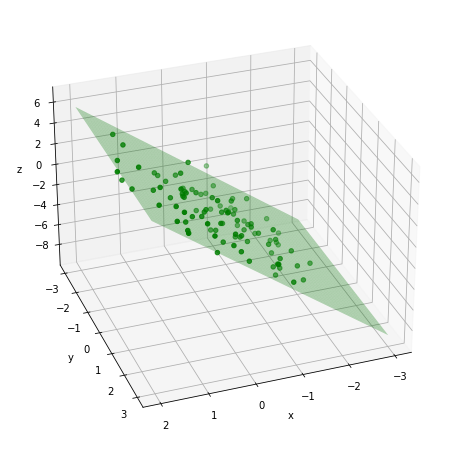
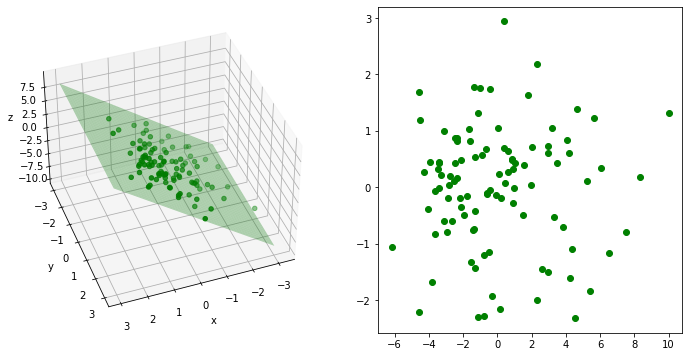
plt.show()可见,在x和y轴上,数据点比较均匀地分布着。但是在z轴方向上,数据点比较集中在一个平面上面。因此降维的一个方式是找到一个平面,把这些数据点投射在这个平面上,将3维数据简化为2维数据。
fig = plt.figure(figsize=(8,8))
ax = fig.add_subplot(1, 1, 1, projection='3d')
ax.scatter(x, y, z, c='g', marker='o')
ax.set_xlabel('x')
ax.set_ylabel('y')
ax.set_zlabel('z')
xgrid = np.linspace(-3, 2, 100)
ygrid = np.linspace(-3, 3, 100)
xgrid, ygrid = np.meshgrid(xgrid, ygrid)
ax.plot_surface(xgrid, ygrid, 3 * xgrid, color = 'green', alpha = 0.3)
ax.view_init(30, 70)流形学习
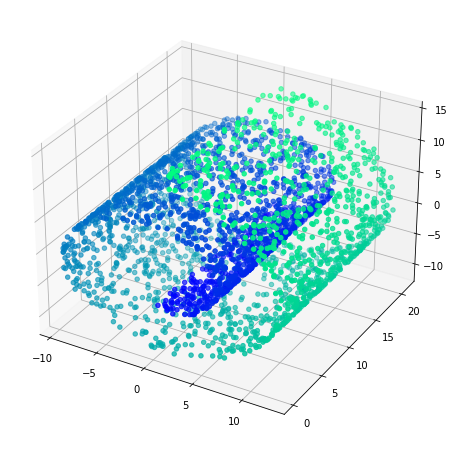
一个$d$维流形可以理解为一个$n$维曲面,其局部可以被$d$维超平面近似。例如,一个在3维空间种的2维流形可以被理解为一个3维曲面,其局部可以被一个2维平面近似。下面的“瑞士卷”是2维流形在3维空间中的例子。
N = 3000
swiss_roll = make_swiss_roll(N)
x, color = swiss_roll
fig = plt.figure(figsize=(8,8))
ax = fig.add_subplot(111, projection='3d')
ax.scatter(x[:,0], x[:,1], x[:,2], c = color, cmap = 'winter')
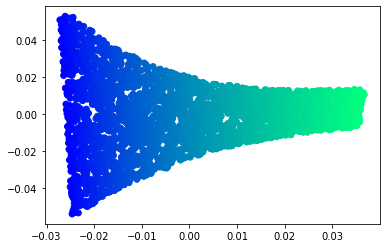
plt.show()如果可以将这个流形展开,我们可以将3维数据集降维到2维。
embedding = LocallyLinearEmbedding(n_components=2, n_neighbors=12)
x_unfold = embedding.fit_transform(x)
fig, ax = plt.subplots()
ax.scatter(x_unfold[:,0], x_unfold[:, 1], c = color, cmap = 'winter')
plt.show()主成分分析(PCA)
主成分分析的思路是投影。核心思想是降维后,保留尽量多的数据方差。步骤是:
- 确定第一个主成分:找到一个向量,使当数据投影到这个向量上面时,方差最大。
- 确定第二个主成分:找到第二个向量与第一个向量正交,使投影到这个向量上的方差最大。
重复这个过程,对于n维数据,最多找到n个主成分。
注意,主成分分解的时候应该先对数据进行转换,使数据集的每个特征的均值为0,主成分向量将默认以0点作为原点。
SVD奇异值分解
设$X$为原数据矩阵,则SVD将其分解为:
$$
\begin{align}
X = U \Sigma V^T
\end{align}
$$
其中,$V$包含了n维矩阵$X$的n个主成分。假设现在要用其中的3个主成分来反映这些数据:
$$
\begin{align}
X_{proj_3} = XW_3
\end{align}
$$
其中,$W_3$包含了$V$的前3列。
下面用SVD法来分解一个类似第一部分的例子。
N = 100
x1 = np.random.normal(0, 1, N)
x2 = np.random.normal(0, 1, N)
x3 = 3 * x1 + np.random.normal(0, 1, N)
xmat = np.stack((x1, x2, x3), axis=1)
xmat.shape
# 均值归0
xmat_centered = xmat - xmat.mean(axis=0) # 对每一个特征去均值
u, sigma, v = svd(xmat_centered)
# 投影:取两个主成分
w2 = v.T[:, :2]
xmat_proj = xmat_centered @ w2
# 画图
fig = plt.figure(figsize=(12,6))
ax1 = fig.add_subplot(1, 2, 1, projection='3d')
ax1.scatter(x1, x2, x3, c='g', marker='o')
ax1.set_xlabel('x')
ax1.set_ylabel('y')
ax1.set_zlabel('z')
ax1.view_init(60, 60)
xgrid = np.linspace(-3, 3, 100)
ygrid = np.linspace(-3, 3, 100)
xgrid, ygrid = np.meshgrid(xgrid, ygrid)
ax1.plot_surface(xgrid, ygrid, 3 * xgrid, color = 'green', alpha = 0.3)
ax1.view_init(40, 70)
ax2 = fig.add_subplot(1, 2, 2)
ax2.scatter(xmat_proj[:,0], xmat_proj[:,1], c = 'g')
plt.show()使用Sklearn包进行SVD
pca = PCA(n_components = 2)
xmat_proj = pca.fit_transform(xmat_centered)方差解释率(Explained Variance Ratio)
某个主成分的方差解释率,是指此主成分保留的方差占原来的总方差的比例。
evr = pca.explained_variance_ratio_
print(evr)[0.89202644 0.09989671]
这意味着,第一个主成分保留了89.2%的方差,第二个主成分保留了10.2%的方差。失去的方差是:
print("{: .2%}".format(1 - evr[0] - evr[1])) 0.81%
选择维度数量
假设要求主成分的方差解释率达到某个值,则在PCA中对n_components中指定一个浮点数。
pca = PCA(n_components=0.95) # 方差解释率达到95%以上
xmat_proj = pca.fit_transform(xmat_centered)
print("Preserve {:n} dimension(s).".format(xmat_proj.shape[1]))Preserve 2 dimension(s).
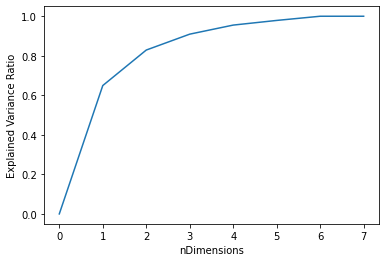
另一方面,我们可以参考维度数量与累计维度解释率的关系。
# 生成示例数据
N = 1000
x1 = np.random.normal(0, 1, N)
x2 = np.random.normal(0, 1, N)
x3 = x1 + x2 * x1 + np.random.normal(0, 1, N)
x4 = x1 + x2**2 + x3 + np.random.normal(0, 1, N)
x5 = x2 + x4 + np.random.normal(0, 1, N)
x6 = x3 + np.random.normal(0, 1, N)
x7 = np.random.normal(0, 1, N)
xmat = np.stack((x1, x2, x3, x4, x5, x6, x7))
# pca
pca = PCA()
pca.fit(xmat) # 不需要去均值;PCA自动做了
cum_evr = np.cumsum(pca.explained_variance_ratio_) # 累计方差解释率
cum_evr = np.insert(cum_evr, 0, 0)
# 画图
fig, ax = plt.subplots()
ax.plot(range(0, len(cum_evr)), cum_evr)
ax.set_xlabel('nDimensions')
ax.set_ylabel('Explained Variance Ratio')
plt.show()